1.4 Manipulation de données
Dans cette section, vous apprendrez à charger et à manipuler des DataFrames en vue d’effectuer des opérations classiques de gestion de données.
1.4.1 Chargement d’un DataFrame depuis un fichier
Il est rarement nécessaire de créer vos DataFrames manuellement. Le plus souvent, vous disposerez de fichiers contenant vos données et utiliserez des fonctions pour les importer dans R sous forme d’un DataFrame. Les formats à importer les plus répandus sont :
- .csv, soit un fichier texte dont chaque ligne représente une ligne du tableau de données et dont les colonnes sont séparées par un délimiteur (généralement une virgule ou un point-virgule);
- .dbf, ou fichier dBase, souvent associés à des fichiers d’information géographique au format ShapeFile;
- .xls et .xlsx, soit des fichiers générés par Excel;
- .json, soit un fichier texte utilisant la norme d’écriture propre au langage JavaScript.
Plus rarement, il se peut que vous ayez à charger des fichiers provenant de logiciels propriétaires :
- .sas7bdat (SAS);
- .sav (SPSS);
- .dta (STATA).
Pour lire la plupart de ces fichiers, nous utilisons le package foreign dédié à l’importation d’une multitude de formats. Nous commençons donc par l’installer (install.packages("foreign")). Ensuite, nous chargeons cinq fois le même jeu de données enregistré dans des formats différents (csv, dbf, dta, sas7bdat et xlsx) et nous mesurons le temps nécessaire pour importer chacun de ces fichiers avec la fonction Sys.time.
1.4.1.1 Lecture d’un fichier csv
Pour le format csv, il n’est pas nécessaire d’utiliser un package puisque R dispose d’une fonction de base pour lire ce format.
t1 <- Sys.time()
df1 <- read.csv("data/priseenmain/SR_MTL_2016.csv",
header = TRUE, sep = ",", dec = ".",
stringsAsFactors = FALSE)
t2 <- Sys.time()
d1 <- as.numeric(difftime(t2,t1,units="secs"))
cat("le DataFrame df1 a ",nrow(df1),' observations',
'et ',ncol(df1),"colonnes\n")## le DataFrame df1 a 951 observations et 48 colonnesRien de bien compliqué! Notez tout de même que :
- Lorsque vous chargez un fichier csv, vous devez connaître le délimiteur (ou séparateur), soit le caractère utilisé pour délimiter les colonnes. Dans le cas présent, il s’agit d’une virgule (spécifiez avec l’argument
sep = ","), mais il pourrait tout aussi bien être un point virgule (sep = ";"), une tabulation (sep = " "), etc. - Vous devez également spécifier le caractère utilisé comme séparateur de décimales. Le plus souvent, ce sera le point (
dec = "."), mais certains logiciels avec des paramètres régionaux de langue française (notamment Excel) exportent des fichiers csv avec des virgules comme séparateur de décimales (utilisez alorsdec = ","). - L’argument
headerindique si la première ligne (l’entête) du fichier comprend ou non les noms des colonnes du jeu de données (avec les valeursTRUEouFALSE). Il arrive que certains fichiers csv soient fournis sans entête et que le nom et la description des colonnes soient fournis dans un autre fichier. - L’argument
stringsAsFactorspermet d’indiquer à R que les colonnes comportant du texte doivent être chargées comme des vecteurs de type texte et non de type facteur.
1.4.1.2 Lecture d’un fichier dbase
Pour lire un fichier dbase (.dbf), nous utilisons la fonction read.dbf du package foreign installé précédemment :
library(foreign)
t1 <- Sys.time()
df2 <- read.dbf("data/priseenmain/SR_MTL_2016.dbf")
t2 <- Sys.time()
d2 <- as.numeric(difftime(t2,t1,units="secs"))
cat("le DataFrame df2 a ",nrow(df2)," observations",
"et ",ncol(df2)," colonnes\n")## le DataFrame df2 a 951 observations et 48 colonnesComme vous pouvez le constater, nous obtenons les mêmes résultats qu’avec le fichier csv.
1.4.1.3 Lecture d’un fichier dta (Stata)
Si vous travaillez avec des collègues utilisant le logiciel Stata, il se peut que ces derniers vous partagent des fichiers dta. Toujours en utilisant le package foreign, vous serez en mesure de les charger directement dans R.
t1 <- Sys.time()
df3 <- read.dta("data/priseenmain/SR_MTL_2016.dta")
t2 <- Sys.time()
d3 <- as.numeric(difftime(t2,t1,units="secs"))
cat("le DataFrame df3 a ",nrow(df3)," observations ",
"et ",ncol(df3),"colonnes\n", sep = "")## le DataFrame df3 a 951 observations et 48colonnes1.4.1.4 Lecture d’un fichier sav (SPSS)
Pour importer un fichier sav provenant du logiciel statistique SPSS, utilisez la fonction read.spss du package foreign.
t1 <- Sys.time()
df4 <- as.data.frame(read.spss("data/priseenmain/SR_MTL_2016.sav"))
t2 <- Sys.time()
d4 <- as.numeric(difftime(t2,t1,units="secs"))
cat("le DataFrame df4 a ",nrow(df4)," observations ",
"et ",ncol(df4),"colonnes\n", sep = "")## le DataFrame df4 a 951 observations et 48colonnes1.4.1.5 Lecture d’un fichier sas7bdat (SAS)
Pour importer un fichier sas7bdat provenant du logiciel statistique SAS, utilisez la fonction read.sas7bdat du package sas7bdat. Installez préalablement le package (install.packages("sas7bdat")) et chargez-le (library(sas7bdat)).
library(sas7bdat)
t1 <- Sys.time()
df5 <- read.sas7bdat("data/priseenmain/SR_MTL_2016.sas7bdat")
t2 <- Sys.time()
d5 <- as.numeric(difftime(t2,t1,units="secs"))
cat("le DataFrame df5 a ",nrow(df5)," observations ",
"et ",ncol(df5)," colonnes\n", sep ="")## le DataFrame df5 a 951 observations et 48 colonnes1.4.1.6 Lecture d’un fichier xlsx (Excel)
Lire un fichier Excel dans R n’est pas toujours une tâche facile. Généralement, nous recommandons d’exporter le fichier en question au format csv dans un premier temps, puis de le lire avec la fonction read.csv dans un second temps (section 1.4.1.1).
Il est néanmoins possible de lire directement un fichier xlsx avec le package xlsx. Ce dernier requiert que le logiciel JAVA soit installé sur votre ordinateur (Windows, Mac ou Linux). Si vous utilisez la version 64 bit de R, vous devrez télécharger et installer la version 64 bit de JAVA. Une fois que ce logiciel tiers est installé, il ne vous restera plus qu’à installer (install.packages("xlsx")) et charger (library(xlsx)) le package xlsx. Sous windows, il est possible que vous deviez également installer manuellement le package rJava et indiquer à R où se trouve JAVA sur votre ordinateur. La procédure est détaillée ici.
library(xlsx)
t1 <- Sys.time()
df6 <- read.xlsx(file="data/priseenmain/SR_MTL_2016.xlsx",
sheetIndex = 1,
as.data.frame = TRUE)
t2 <- Sys.time()
d6 <- as.numeric(difftime(t2,t1,units="secs"))
cat("le DataFrame df6 a ",nrow(df6)," observations ",
"et ",ncol(df6)," colonnes\n", sep = "")## le DataFrame df6 a 951 observations et 48 colonnesIl est possible d’accélérer significativement la vitesse de lecture d’un fichier xlsx en utilisant la fonction read.xlsx2. Il faut cependant indiquer à cette dernière le type de données de chaque colonne. Dans le cas présent, les cinq premières colonnes contiennent des données de type texte (character), alors que les 43 autres sont des données numériques (numeric). Nous utilisons la fonction rep afin de ne pas avoir à écrire plusieurs fois character et numeric.
library(xlsx)
t1 <- Sys.time()
df7 <- read.xlsx2(file="data/priseenmain/SR_MTL_2016.xlsx",
sheetIndex = 1,
as.data.frame = TRUE,
colClasses = c(rep("character",5),rep("numeric",43))
)
t2 <- Sys.time()
d7 <- as.numeric(difftime(t2,t1,units="secs"))
cat("le DataFrame df6 a ",nrow(df7)," observations ",
"et ",ncol(df7),"colonnes\n", sep = "")## le DataFrame df6 a 951 observations et 48colonnesSi nous comparons les temps d’exécution (tableau 1.5), nous constatons que la lecture des fichiers xlsx peut être extrêmement longue si nous ne spécifions pas le type des colonnes, ce qui peut devenir problématique pour des fichiers volumineux. Notez également que la lecture d’un fichier csv devient de plus en plus laborieuse à mesure que sa taille augmente. Si vous devez un jour charger des fichiers csv de plusieurs gigaoctets, nous vous recommandons vivement d’utiliser la fonction fread du package data.table qui est beaucoup plus rapide.
| Durée (secondes) | Fonction |
|---|---|
| 0,04 | read.csv |
| 0,02 | read.dbf |
| 0,00 | read.spss |
| 0,01 | read.dta |
| 0,92 | read.sas7bdat |
| 13,77 | read.xlsx |
| 0,23 | read.xlsx2 |
1.4.2 Manipulation d’un DataFrame
Une fois le DataFrame chargé, voyons comment il est possible de le manipuler.
1.4.2.1 Petit mot sur le tidyverse
tidyverse est un ensemble de packages conçus pour faciliter la structuration et la manipulation des données dans R. Avant d’aller plus loin, il est important d’aborder brièvement un débat actuel dans la Communauté R. Entre 2010 et 2020, l’utilisation du tidyverse s’est peu à peu répandue. Développé et maintenu par Hadley Wickham, tidyverse introduit une philosophie et une grammaire spécifiques qui diffèrent du langage R traditionnel. Une partie de la communauté a pour ainsi dire complètement embrassé le tidyverse et de nombreux packages, en dehors du tidyverse, ont adopté sa grammaire et sa philosophie. À l’inverse, une autre partie de la communauté est contre cette évolution (voir l’article du blogue suivant). Les arguments pour et contre tidyverse sont résumés dans le tableau suivant.
| Avantage du tidyverse | Problème posé par le tidyverse |
|---|---|
| Simplicité d’écriture et d’apprentissage | Nouvelle syntaxe à apprendre |
Ajout de l’opérateur %>% permettant d’enchaîner les traitements
|
Perte de lisibilité avec l’opérateur ->
|
La meilleure librairie pour réaliser des graphiques : ggplot2
|
Remplacement de certaines fonctions de base par d’autres provenant du tidyverse lors de son chargement, pouvant créer des erreurs.
|
| Crée un écosystème cohérent | Ajout d’une dépendance dans le code |
| Package en développement et de plus en plus utilisé | Philosophie d’évolution agressive, aucune assurance de rétrocompatibilité |
Le dernier point est probablement le plus problématique. Dans sa volonté d’évoluer au mieux et sans restriction, le package tidyverse n’offre aucune garantie de rétrocompatibilité. En d’autre termes, des changements importants peuvent être introduits d’une version à l’autre rendant potentiellement obsolète votre ancien code. Nous n’avons pas d’opinion tranchée sur le sujet : tidyverse est un outil très intéressant dans de nombreux cas; nous évitons simplement de l’utiliser systématiquement et préférons charger directement des sous-packages (comme dplyr ou ggplot2) du tidyverse. Notez que le package data.table offre une alternative au tidyverse dans la manipulation de données. Au prix d’une syntaxe généralement un peu plus complexe, le package data.table offre une vitesse de calcul bien supérieure au tidyverse et assure une bonne rétrocompatibilité.
1.4.2.2 Gestion des colonnes d’un DataFrame
Repartons du DataFrame que nous avions chargé précédemment en important un fichier csv.
df <- read.csv(file="data/priseenmain/SR_MTL_2016.csv",
header = TRUE, sep = ",", dec = ".",
stringsAsFactors = FALSE)1.4.2.2.1 Sélection d’une colonne
Rappelons qu’il est possible d’accéder aux colonnes dans ce DataFrame en utilisant le symbole dollar $ma_colonne ou les doubles crochets [["ma_colonne"]].
# Calcul de la superficie totale de l'Île de Montréal
sum(df$KM2)## [1] 4680.543sum(df[["KM2"]])## [1] 4680.5431.4.2.2.2 Sélection de plusieurs colonnes
Il est possible de sélectionner plusieurs colonnes d’un DataFrame et de filtrer ainsi les colonnes inutiles. Pour cela, nous pouvons utiliser un vecteur contenant soit les positions des colonnes (1 pour la première colonne, 2 pour la seconde et ainsi de suite), soit les noms des colonnes.
# Conserver les 5 premières colonnes
df2 <- df[1:5]
# Conserver les colonnes 1, 5, 10 et 15
df3 <- df[c(1,5,10,15)]
# Cela peut aussi être utilisé pour changer l'ordre des champs
df3 <- df[c(10,15,1,5)]
# Conserver les colonnes 1 à 5, 7 à 12, 17 et 22
df4 <- df[c(1:5,7:12,17,22)]
# Conserver les colonnes avec leurs noms
df5 <- df[c("SRIDU","KM2","Pop2016","MaisonIndi","LoyerMed")]1.4.2.2.3 Suppression de colonnes
Il est parfois plus intéressant et rapide de supprimer directement des colonnes plutôt que de recréer un nouveau DataFrame. Pour ce faire, nous attribuons la valeur NULL à ces colonnes.
# Supprimer le colonnes 2, 3 et 5
df3[c(2,3,5)] <- list(NULL)
# Supprimer une colonne avec son nom
df4$OID <- NULL
# Supprimer plusieurs colonnes par leur nom
df5[c("SRIDU","LoyerMed")] <- list(NULL)Notez que si vous supprimez une colonne, vous ne pouvez pas revenir en arrière. Il faudra recharger votre jeu de données ou éventuellement relancer les calculs qui avaient produit cette colonne.
1.4.2.2.4 Modification du nom des colonnes
Il est possible de changer le nom d’une colonne. Cette opération est importante pour faciliter la lecture du DataFrame ou encore s’assurer que l’exportation du DataFrame dans un format particulier (tel que .dbf qui ne supporte que les noms de colonnes avec moins de 10 caractères) ne posera pas de problème.
# Voici les noms des colonnes
names(df5)## [1] "KM2" "Pop2016" "MaisonIndi"# Renommer toutes les colonnes
names(df5) <- c('superficie_km2','population_2016', 'maison_individuelle_prt')
names(df5)## [1] "superficie_km2" "population_2016" "maison_individuelle_prt"# Renommer avec dplyr
library(dplyr)
df4 <- rename(df4, "population_2016" = "Pop2016",
"prs_moins_14ans_prt" = "A014",
"prs_15_64_ans_prt" = "A1564",
"prs_65plus_ans_prt" = "A65plus"
)1.4.2.3 Calcul de nouvelles variables
Il est possible d’utiliser les colonnes de type numérique pour calculer de nouvelles colonnes en utilisant les opérateurs mathématiques vus dans la section 1.3.5. Prenons un exemple concret : calculons la densité de population par secteur de recensement dans notre DataFrame, puis affichons un résumé de cette nouvelle variable.
# Calcul de la densité
df$pop_density_2016 <- df$Pop2016 / df$KM2
# Statistiques descriptives
summary(df$pop_density_2016)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 17.45 1946.96 3700.50 5465.03 7918.39 48811.79Nous pouvons aussi calculer le ratio entre le nombre de maisons et le nombre d’appartements.
# Calcul du ratio
df$total_maison <- (df$MaisonIndi + df$MaisJumule + df$MaisRangee + df$AutreMais)
df$total_apt <- (df$AppDuplex + df$App5Moins + df$App5Plus)
df$ratio_maison_apt <- df$total_maison / df$total_aptRetenez ici que R applique le calcul à chaque ligne de votre jeu de données et stocke le résultat dans une nouvelle colonne. Cette opération est du calcul vectoriel : toute la colonne est calculée en une seule fois. R est d’ailleurs optimisé pour le calcul vectoriel.
1.4.2.4 Fonctions mathématiques
R propose un ensemble de fonctions de base pour effectuer du calcul. Voici une liste non exhaustive des principales fonctions :
abscalcule la valeur absolue de chaque valeur d’un vecteur;sqrtcalcule la racine carrée de chaque valeur d’un vecteur;logcalcule le logarithme de chaque valeur d’un vecteur;expcalcule l’exponentielle de chaque valeur d’un vecteur;factorialcalcule la factorielle de chaque valeur d’un vecteur;roundarrondit la valeur d’un vecteur;ceiling,floorarrondit à l’unité supérieure ou inférieure de chaque valeur d’un vecteur;sin,asin,cos,acos,tan,atansont des fonctions de trigonométrie;cumsumcalcule la somme cumulative des valeurs d’un vecteur.
Ces fonctions sont des fonctions vectorielles puisqu’elles s’appliquent à tous les éléments d’un vecteur. Si votre vecteur en entrée comprend cinq valeurs, le vecteur en sortie comprendra aussi cinq valeurs.
À l’inverse, les fonctions suivantes s’appliquent directement à l’ensemble d’un vecteur et ne vont renvoyer qu’une seule valeur :
sumcalcule la somme des valeurs d’un vecteur;prodcalcule le produit des valeurs d’un vecteur;min,maxrenvoient les valeurs maximale et minimale d’un vecteur;mean,medianrenvoient la moyenne et la médiane d’un vecteur;quantilerenvoie les percentiles d’un vecteur.
1.4.2.5 Fonctions pour manipuler des chaînes de caractères
Outre les données numériques, vous aurez à travailler avec des données de type texte (string). Le tidyverse avec le package stringr offre des fonctions très intéressantes pour manipuler ce type de données. Pour un aperçu de toutes les fonctions offertes par stringr, référez-vous à sa Cheat Sheet. Commençons avec un DataFrame assez simple comprenant des adresses et des noms de personnes.
library(stringr)
df <- data.frame(
noms = c("Jérémy Toutanplace","constant Tinople","dino Resto","Luce tancil"),
adresses = c('15 rue Levy', '413 Blvd Saint-Laurent', '3606 rue Duké', '2457 route St Marys')
)1.4.2.5.1 Majuscules et minuscules
Pour harmoniser ce DataFrame, nous mettons, dans un premier temps, des majuscules à la première lettre des prénoms et des noms des individus avec la fonction str_to_title.
df$noms_corr <- str_to_title(df$noms)
print(df$noms_corr)## [1] "Jérémy Toutanplace" "Constant Tinople" "Dino Resto" "Luce Tancil"Nous pourrions également tout mettre en minuscules ou tout en majuscules.
df$noms_min <- tolower(df$noms)
df$noms_maj <- toupper(df$noms)
print(df$noms_min)## [1] "jérémy toutanplace" "constant tinople" "dino resto" "luce tancil"print(df$noms_maj)## [1] "JÉRÉMY TOUTANPLACE" "CONSTANT TINOPLE" "DINO RESTO" "LUCE TANCIL"1.4.2.5.2 Remplacement du texte
Les adresses comprennent des caractères accentués. Ce type de caractères cause régulièrement des problèmes d’encodage. Nous pourrions alors décider de les remplacer par des caractères simples avec la fonction str_replace_all.
df$adresses_1 <- str_replace_all(df$adresses,'é','e')
print(df$adresses_1)## [1] "15 rue Levy" "413 Blvd Saint-Laurent" "3606 rue Duke" "2457 route St Marys"La même fonction peut être utilisée pour remplacer les St par Saint et les Blvd par Boulevard.
df$adresses_2 <- str_replace_all(df$adresses_1,' St ',' Saint ')
df$adresses_3 <- str_replace_all(df$adresses_2,' Blvd ',' Boulevard ')
print(df$adresses_3)## [1] "15 rue Levy" "413 Boulevard Saint-Laurent" "3606 rue Duke"
## [4] "2457 route Saint Marys"1.4.2.5.3 Découpage du texte
Il est parfois nécessaire de découper du texte pour en extraire des éléments. Nous devons alors choisir un caractère de découpage. Dans notre exemple, nous pourrions vouloir extraire les numéros civiques des adresses en sélectionnant le premier espace comme caractère de découpage, et en utilisant la fonction str_split_fixed.
df$num_civique <- str_split_fixed(df$adresses_3, ' ',n=2)[,1]
print(df$num_civique)## [1] "15" "413" "3606" "2457"Pour être exact, sachez que pour notre exemple, la fonction str_split_fixed renvoie deux colonnes de texte : une avec le texte avant le premier espace, soit le numéro civique, et une avec le reste du texte. Le nombre de colonnes est contrôlé par l’argument n. Si n = 1, la fonction ne fait aucun découpage; avec n = 2 la fonction découpe en deux parties le texte avec la première occurrence du délimiteur et ainsi de suite. En ajoutant [,1] à la fin, nous indiquons que nous souhaitons garder seulement la première des deux colonnes.
Il est également possible d’extraire des parties de texte et de ne garder par exemple que les N premiers caractères ou les N derniers caractères :
# Ne garder que les 5 premiers caractères
substr(df$adresses_3,start = 1, stop = 5)## [1] "15 ru" "413 B" "3606 " "2457 "# Ne garder que les 5 derniers caractères
n_caract <- nchar(df$adresses_3)
substr(df$adresses_3, start = n_caract-4, stop = n_caract)## [1] " Levy" "urent" " Duke" "Marys"Notez que les paramètres start et stop de la fonction substr peuvent accepter un vecteur de valeurs. Il est ainsi possible d’appliquer une sélection de texte différente à chaque chaîne de caractères dans notre vecteur en entrée. Nous pourrions par exemple vouloir récupérer tout le texte avant le second espace pour garder uniquement le numéro civique et le type de rue.
# Étape 1 : récupérer les positions des espaces pour chaque adresses
positions <- str_locate_all(df$adresses_3, " ")
# Étape 2 : récupérer les positions des seconds espaces
sec_positions <- sapply(positions, function(i){
i[2,1]
})
# Étape 3 : appliquer le découpage
substr(df$adresses_3, start = 1, stop = sec_positions-1)## [1] "15 rue" "413 Boulevard" "3606 rue" "2457 route"1.4.2.5.4 Concaténation du texte
À l’inverse du découpage, il est parfois nécessaire de concaténer des éléments de texte, ce qu’il est possible de réaliser avec la fonction paste.
df$texte_complet <- paste(df$noms_corr, df$adresses_3, sep = " : ")
print(df$texte_complet)## [1] "Jérémy Toutanplace : 15 rue Levy" "Constant Tinople : 413 Boulevard Saint-Laurent"
## [3] "Dino Resto : 3606 rue Duke" "Luce Tancil : 2457 route Saint Marys"Le paramètre sep permet d’indiquer le ou les caractères à intercaler entre les éléments à concaténer. Notez qu’il est possible de concaténer plus que deux éléments.
df$ville <- c('Montreal','Montreal','Montreal','Montreal')
paste(df$noms_corr, df$adresses_3, df$ville, sep = ", ")## [1] "Jérémy Toutanplace, 15 rue Levy, Montreal"
## [2] "Constant Tinople, 413 Boulevard Saint-Laurent, Montreal"
## [3] "Dino Resto, 3606 rue Duke, Montreal"
## [4] "Luce Tancil, 2457 route Saint Marys, Montreal"Si vous souhaitez concaténer des éléments de texte sans séparateur, la fonction paste0 peut être plus simple à utiliser.
paste0("Please conca", "tenate me!")## [1] "Please concatenate me!"1.4.2.6 Manipulation des colonnes de type date
Nous avons vu que les principaux types de données dans R sont le numérique, le texte, le booléen et le facteur. Il existe d’autres types introduits par différents packages. Nous abordons ici les types date et heure (date and time). Pour les manipuler, nous privilégions l’utilisation du package lubridate du tidyverse. Pour illustrer le tout, nous l’utilisons avec un jeu de données ouvertes de la Ville de Montréal représentant les collisions routières impliquant au moins un cycliste survenues après le 1er janvier 2017.
accidents_df <- read.csv(file="data/priseenmain/accidents.csv", sep = ",")
names(accidents_df)## [1] "HEURE_ACCDN" "DT_ACCDN" "NB_VICTIMES_TOTAL"Nous disposons de trois colonnes représentant respectivement l’heure, la date et le nombre de victimes impliquées dans la collision.
1.4.2.6.1 Du texte à la date
Actuellement, les colonnes HEURE_ACCDN et DT_ACCDN sont au format texte. Nous pouvons afficher quelques lignes du jeu de données avec la fonction head pour visualiser comment elles ont été saisies.
head(accidents_df, n = 5)## HEURE_ACCDN DT_ACCDN NB_VICTIMES_TOTAL
## 1 16:00:00-16:59:00 2017/11/02 0
## 2 06:00:00-06:59:00 2017/01/16 1
## 3 18:00:00-18:59:00 2017/04/18 0
## 4 11:00:00-11:59:00 2017/05/28 1
## 5 15:00:00-15:59:00 2017/05/28 1Un peu de ménage s’impose : les heures sont indiquées comme des périodes d’une heure. Nous utilisons la fonction str_split_fixed du package stringr pour ne garder que la première partie de l’heure (avant le tiret). Ensuite, Nous concaténons l’heure et la date avec la fonction paste, puis nous convertissons ce résultat en un objet date-time.
library(lubridate)
# Étape 1 : découper la colonne Heure_ACCDN
accidents_df$heure <- str_split_fixed(accidents_df$HEURE_ACCDN, "-", n=2)[,1]
# Étape 2 : concaténer l'heure et la date
accidents_df$date_heure <- paste(accidents_df$DT_ACCDN,
accidents_df$heure,
sep = ' ')
# Étape 3 : convertir au format datetime
accidents_df$datetime <- as_datetime(accidents_df$date_heure,
format = "%Y/%m/%d %H:%M:%S")Pour effectuer la conversion, nous avons utilisé la fonction as_datetime du package lubridate. Elle prend comme paramètre un vecteur de texte et une indication du format de ce vecteur de texte. Il existe de nombreuses façons de spécifier une date et une heure et l’argument format permet d’indiquer celle à utiliser. Dans cet exemple, la date est structurée comme suit :
année/mois/jour heure:minute:seconde, ce qui se traduit par le format %Y/%m/%d %H:%M:%S.
- %Y signifie une année indiquée avec quatre caractères : 2017;
- %m signifie un mois, indiqué avec deux caractères : 01, 02, 03, … 12;
- %d signifie un jour, indiqué avec deux caractères : 01, 02, 03, … 31;
- %H signifie une heure, au format 24 heures avec deux caractères : 00, 02, … 23;
- %M signifie des minutes indiquées avec deux caractères : 00, 02, … 59;
- %S signifie des secondes, indiquées avec deux caractères : 00, 02, … 59.
Notez que les caractères séparant les années, jours, heures, etc. sont aussi à indiquer dans le format. Dans notre exemple, nous utilisons la barre oblique (/) pour séparer les éléments de la date et le deux points (:) pour l’heure, et une espace pour séparer la date et l’heure.
Il existe d’autres nomenclatures pour spécifier un format datetime : par exemple, des mois renseignés par leur nom, l’indication AM-PM, etc. Vous pouvez vous référer à la documentation de la fonction strptime (help(strptime)) pour explorer les différentes nomenclatures et choisir celle qui vous convient. Bien évidemment, il est nécessaire que toutes les dates de votre colonne soient renseignées dans le même format, pour éviter que la fonction ne retourne la valeur NA lorsqu’elle ne peut lire le format. Après toutes ces opérations, rejetons un oeil à notre DataFrame.
head(accidents_df, n = 5)## HEURE_ACCDN DT_ACCDN NB_VICTIMES_TOTAL heure date_heure datetime
## 1 16:00:00-16:59:00 2017/11/02 0 16:00:00 2017/11/02 16:00:00 2017-11-02 16:00:00
## 2 06:00:00-06:59:00 2017/01/16 1 06:00:00 2017/01/16 06:00:00 2017-01-16 06:00:00
## 3 18:00:00-18:59:00 2017/04/18 0 18:00:00 2017/04/18 18:00:00 2017-04-18 18:00:00
## 4 11:00:00-11:59:00 2017/05/28 1 11:00:00 2017/05/28 11:00:00 2017-05-28 11:00:00
## 5 15:00:00-15:59:00 2017/05/28 1 15:00:00 2017/05/28 15:00:00 2017-05-28 15:00:001.4.2.6.2 Extraction des informations d’une date
À partir de la nouvelle colonne datetime, nous sommes en mesure d’extraire des informations intéressantes comme :
- le nom du jour de la semaine avec la fonction
weekdays
accidents_df$jour <- weekdays(accidents_df$datetime)- la période de la journée avec les fonctions
ametpm
accidents_df$AM <- am(accidents_df$datetime)
accidents_df$PM <- pm(accidents_df$datetime)
head(accidents_df[c("jour", "AM", "PM")], n=5)## jour AM PM
## 1 jeudi FALSE TRUE
## 2 lundi TRUE FALSE
## 3 mardi FALSE TRUE
## 4 dimanche TRUE FALSE
## 5 dimanche FALSE TRUEIl est aussi possible d’accéder aux sous-éléments d’un datetime comme l’année, le mois, le jour, l’heure, la minute et la seconde avec les fonctions year(), month(),day(), hour(), minute() et second().
1.4.2.6.3 Calcul d’une durée entre deux datetime
Une autre utilisation intéressante du format datetime est de calculer des différences de temps. Par exemple, nous pourrions utiliser le nombre de minutes écoulées depuis 7 h comme une variable dans une analyse visant à déterminer le moment critique des collisions routières durant l’heure de pointe du matin.
Pour cela, nous devons créer un datetime de référence en concaténant la date de chaque observation, et le temps 07:00:00, qui est notre point de départ.
accidents_df$date_heure_07 <- paste(accidents_df$DT_ACCDN,
'07:00:00',
sep = ' ')
accidents_df$ref_datetime <- as_datetime(accidents_df$date_heure_07,
format = "%Y/%m/%d %H:%M:%S")Il ne nous reste plus qu’à calculer la différence de temps entre la colonne datetime et notre temps de référence ref_datetime.
accidents_df$diff_time <- difftime(accidents_df$datetime,
accidents_df$ref_datetime,
units = 'min')Notez qu’ici la colonne diff_time est d’un type spécial : une différence temporelle (difftime). Il faut encore la convertir au format numérique pour l’utiliser avec la fonction as.numeric. Par curiosité, réalisons rapidement un histogramme avec la fonction hist pour analyser rapidement cette variable d’écart de temps!
accidents_df$diff_time_num <- as.numeric(accidents_df$diff_time)
hist(accidents_df$diff_time_num, breaks = 50)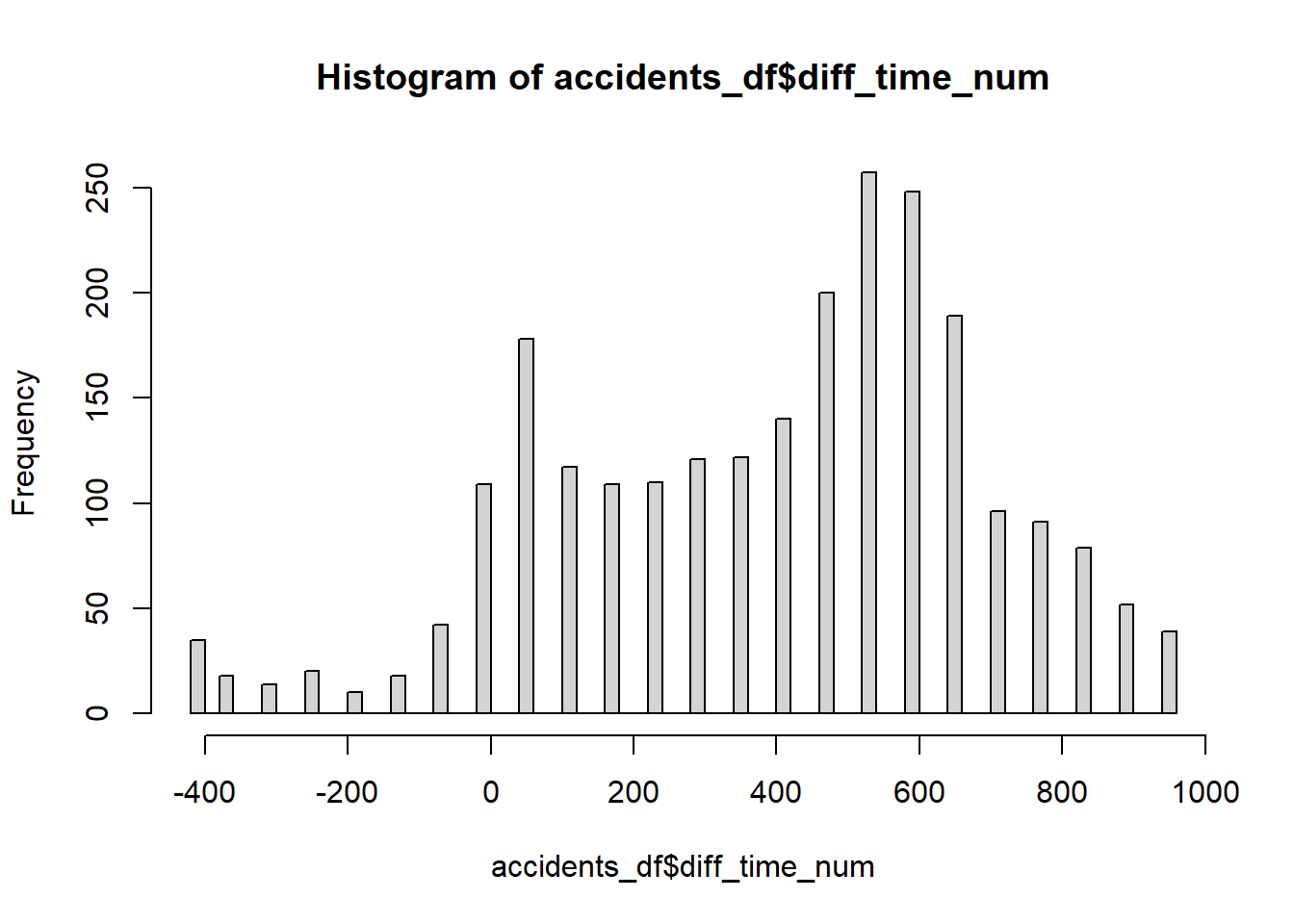
Figure 1.15: Répartition temporelle des accidents à vélo
Nous observons clairement deux pics, un premier entre 0 et 100 (entre 7 h et 8 h 30 environ) et un second plus important entre 550 et 650 (entre 16 h et 17 h 30 environ), ce qui correspond sans surprise aux heures de pointe (figure 1.15). Il est intéressant de noter que plus d’accidents se produisent à l’heure de pointe du soir qu’à celle du matin.
1.4.2.6.4 Fuseau horaire
Lorsque nous travaillons avec des données provenant de différents endroits dans le monde ou que nous devons tenir compte des heures d’été et d’hiver, il convient de tenir compte du fuseau horaire. Pour créer une date avec un fuseau horaire, il est possible d’utiliser le paramètre tz dans la fonction as_datetime et d’utiliser l’identifiant du fuseau approprié. Dans notre cas, les données d’accident ont été collectées à Montréal, qui a un décalage de -5 heures par rapport au temps de référence UTC (+1 heure en été). Le code spécifique de ce fuseau horaire est EDT; il est facile de trouver ces codes avec le site web timeanddate.com.
accidents_df$datetime <- as_datetime(accidents_df$date_heure,
format = "%Y/%m/%d %H:%M:%S",
tz = "EDT")1.4.2.7 Recodage des variables
Recoder une variable signifie changer ses valeurs selon une condition afin d’obtenir une nouvelle variable. Si nous reprenons le jeu de données précédent sur les accidents à vélo, nous pourrions vouloir créer une nouvelle colonne nous indiquant si la collision a eu lieu en heures de pointe ou non. Nous obtiendrions ainsi une nouvelle variable avec seulement deux catégories plutôt que la variable numérique originale. Nous pourrions aussi définir quatre catégories avec l’heure de pointe du matin, l’heure de pointe du soir, le reste de la journée et la nuit.
1.4.2.7.1 Cas binaire avec ifelse
Si nous ne souhaitons créer que deux catégories, le plus simple est d’utiliser la fonction ifelse. Cette fonction évalue une condition (section 1.3.5) pour chaque ligne d’un DataFrame et produit un nouveau vecteur. Créons donc une variable binaire indiquant si une collision a eu lieu durant les heures de pointe ou hors heures de pointe. Nous devons alors évaluer les conditions suivantes :
Est-ce que l’accident a eu lieu entre 7 h (0) ET 9 h (120), OU entre 16 h 30 (570) ET 18 h 30 (690)?
table(is.na(accidents_df$diff_time_num))##
## FALSE TRUE
## 2414 40Notons dans un premier temps que nous avons 40 observations sans valeur pour la colonne diff_time_num. Il s’agit d’observations pour lesquelles nous ne disposions pas de dates au départ.
Cond1 <- accidents_df$diff_time_num >= 0 & accidents_df$diff_time_num <= 120
Cond2 <- accidents_df$diff_time_num >= 570 & accidents_df$diff_time_num <= 690
accidents_df$moment_bin <- ifelse(Cond1 | Cond2,
"en heures de pointe",
"hors heures de pointe")Comme vous pouvez le constater, la fonction ifelse nécessite trois arguments :
- une condition, pouvant être
TRUEouFALSE; - la valeur à renvoyer si la condition est
FALSE; - la valeur à renvoyer si la condition est
TRUE.
Avec la fonction table, nous pouvons rapidement visualiser les effectifs des deux catégories ainsi créées :
table(accidents_df$moment_bin)##
## en heures de pointe hors heures de pointe
## 841 1573# Vérifier si nous avons toujours seulement 40 NA
table(is.na(accidents_df$moment_bin))##
## FALSE TRUE
## 2414 40Les heures de pointe représentent quatre heures de la journée, ce qui nous laisse neuf heures hors heures de pointe entre 7 h et 20 h.
# Ratio de collisions routières en heures de pointe
(841 / 2414) / (4 / 13)## [1] 1.132249# Ratio de collisions routières hors heure de pointe
(1573 / 2414) / (9 / 13)## [1] 0.9412225En rapportant les collisions aux durées des deux périodes, nous observons une nette surreprésentation des collisions impliquant un vélo pendant les heures de pointe d’environ 13 % comparativement à la période hors des heures de pointe.
1.4.2.7.2 Cas multiple avec la case_when
Lorsque nous souhaitons créer plus que deux catégories, il est possible soit d’enchaîner plusieurs fonctions ifelse (ce qui produit un code plus long et moins lisible), soit d’utiliser la fonction case_when du package dplyr du tidyverse. Reprenons notre exemple et créons quatre catégories :
- en heures de pointe du matin;
- en heures de pointe du soir;
- le reste de la journée (entre 7 h et 20 h);
- la nuit (entre 21 h et 7 h).
library(dplyr)
accidents_df$moment_multi <- case_when(
accidents_df$diff_time_num >= 0 & accidents_df$diff_time_num <= 120 ~ "pointe matin",
accidents_df$diff_time_num >= 570 & accidents_df$diff_time_num <= 690 ~ "pointe soir",
accidents_df$diff_time_num > 690 & accidents_df$diff_time_num < 780 ~ "journee",
accidents_df$diff_time_num > 120 & accidents_df$diff_time_num < 570 ~ "journee",
accidents_df$diff_time_num < 0 | accidents_df$diff_time_num >= 780 ~ "nuit"
)
table(accidents_df$moment_multi)##
## journee nuit pointe matin pointe soir
## 1155 418 404 437# Vérifions encore les NA
table(is.na(accidents_df$moment_multi))##
## FALSE TRUE
## 2414 40La syntaxe de cette fonction est un peu particulière. Elle accepte un nombre illimité (ou presque) d’arguments. Chaque argument est composé d’une condition et d’une valeur à renvoyer si la condition est vraie; ces deux éléments étant reliés par le symbole ~. Notez que toutes les évaluations sont effectuées dans l’ordre des arguments. En d’autres termes, la fonction teste d’abord la première condition et assigne ses valeurs, puis recommence pour les prochaines conditions. Ainsi, si une observation (ligne du tableau de données) obtient TRUE à plusieurs conditions, elle obtient au final la valeur de la dernière condition validée. Dans l’exemple précédent, si la première condition est accidents_df$diff_time_num >= 0 | accidents_df$diff_time_num <= 120, alors nous obtenons pour seule valeur en résultat "pointe matin" puisque chaque observation a une valeur supérieure à 0 et que nous avons remplacé l’opérateur & (ET) par l’opérateur | (OU).
1.4.2.8 Sous-sélection d’un DataFrame
Dans cette section, nous voyons comment extraire des sous-parties d’un DataFrame. Il est possible de sous-sélectionner des lignes et des colonnes en se basant sur des conditions ou leur index. Pour cela, nous utilisons un jeu de données fourni avec R : le jeu de données iris décrivant des fleurs du même nom.
data("iris")
# Nombre de lignes et de colonnes
dim(iris)## [1] 150 51.4.2.8.1 Sous-sélection des lignes
Sous-sélectionner des lignes par index est relativement simple. Admettons que nous souhaitons sélectionner les lignes 1 à 5, 10 à 25, 37 et 58.
sub_iris <- iris[c(1:5, 10:25, 37, 58),]
nrow(sub_iris)## [1] 23Sous-sélectionner des lignes avec une condition peut être effectué soit avec une syntaxe similaire, soit en utilisant la fonction subset. Sélectionnons toutes les fleurs de l’espèce Virginica.
iris_virginica1 <- iris[iris$Species == "virginica",]
iris_virginica2 <- subset(iris, iris$Species == "virginica")
# Vérifions que les deux DataFrames ont le même nombre de lignes
nrow(iris_virginica1) == nrow(iris_virginica2)## [1] TRUEVous pouvez utiliser, dans les deux cas, tous les opérateurs vus dans les sections 1.3.5.2 et 1.3.5.3. L’enjeu est d’arriver à créer un vecteur booléen final permettant d’identifier les observations à conserver.
1.4.2.8.2 Sous-sélection des colonnes
Nous avons déjà vu comment sélectionner des colonnes en utilisant leur nom ou leur index dans la section 1.4.2.2.1. Ajoutons ici un cas particulier où nous souhaitons sélectionner des colonnes selon une condition. Par exemple, nous pourrions vouloir conserver que les colonnes comprenant le mot Length. Pour cela, nous utilisons la fonction grepl, permettant de déterminer si des caractères sont présents dans une chaîne de caractères.
nom_cols <- names(iris)
print(nom_cols)## [1] "Sepal.Length" "Sepal.Width" "Petal.Length" "Petal.Width" "Species"test_nom <- grepl("Length",nom_cols, fixed = TRUE)
ok_nom <- nom_cols[test_nom]
iris_2 <- iris[ok_nom]
print(names(iris_2))## [1] "Sepal.Length" "Petal.Length"Il est possible d’obtenir ce résultat en une seule ligne de code, mais elle est un peu moins lisible.
iris2 <- iris[names(iris)[grepl("Length",names(iris), fixed = TRUE)]]1.4.2.8.3 Sélection des colonnes et des lignes
Nous avons vu qu’avec les crochets [], nous pouvons extraire les colonnes et les lignes d’un DataFrame. Il est possible de combiner les deux opérations simultanément. Pour ce faire, il faut indiquer en premier les index ou la condition permettant de sélectionner une ligne, puis les index ou la condition pour sélectionner les colonnes : [index_lignes , index_colonnes]. Sélectionnons cinq premières lignes et les trois premières colonnes du jeu de données iris :
iris_5x3 <- iris[c(1,2,3,4,5),c(1,2,3)]
print(iris_5x3)## Sepal.Length Sepal.Width Petal.Length
## 1 5.1 3.5 1.4
## 2 4.9 3.0 1.4
## 3 4.7 3.2 1.3
## 4 4.6 3.1 1.5
## 5 5.0 3.6 1.4Combinons nos deux exemples précédents pour sélectionner uniquement les lignes avec des fleurs de l’espèce virginica, et les colonnes avec le mot Length.
iris_virginica3 <- iris[iris$Species == "virginica",
names(iris)[grepl("Length",names(iris), fixed = TRUE)]]
head(iris_virginica3, n=5)## Sepal.Length Petal.Length
## 101 6.3 6.0
## 102 5.8 5.1
## 103 7.1 5.9
## 104 6.3 5.6
## 105 6.5 5.81.4.2.9 Fusion de DataFrames
Terminons cette section avec la fusion de DataFrames. Nous distinguons deux méthodes répondant à des besoins différents : par ajout ou par jointure.
1.4.2.9.1 Fusion de DataFrames par ajout
Ajouter deux DataFrames peut se faire en fonction de leurs colonnes ou en fonction de leurs lignes. Dans ces deux cas, nous utilisons respectivement les fonctions cbind et rbind. La figure 1.16 résume graphiquement le fonctionnement des deux fonctions.
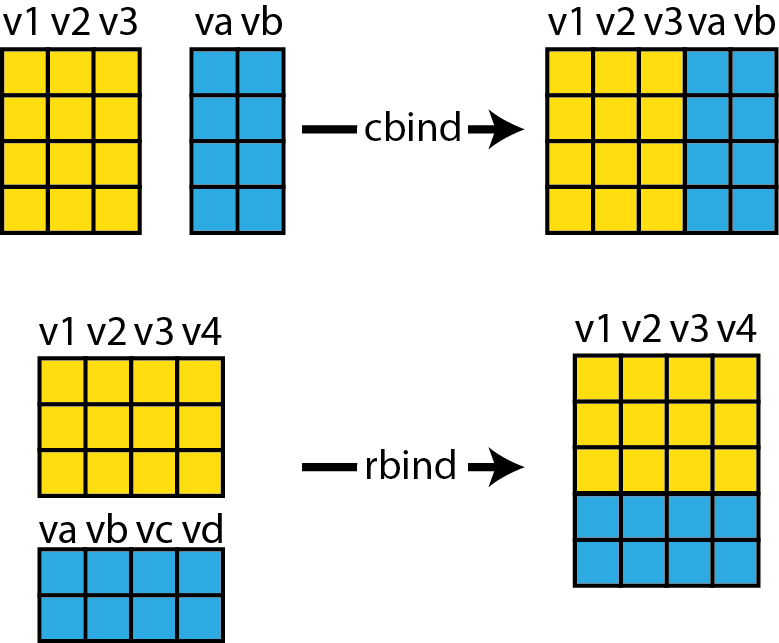
Figure 1.16: Fusion de DataFrames
Pour que cbind fonctionne, il faut que les deux DataFrames aient le même nombre de lignes. Pour rbind, les deux DataFrames doivent avoir le même nombre de colonnes. Prenons à nouveau comme exemple le jeu de données iris. Nous commençons par le séparer en trois sous-jeux de données comprenant chacun une espèce d’iris. Puis, nous fusionnons deux d’entre eux avec la fonction rbind.
iris1 <- subset(iris, iris$Species == "virginica")
iris2 <- subset(iris, iris$Species == "versicolor")
iris3 <- subset(iris, iris$Species == "setosa")
iris_comb <- rbind(iris2,iris3)Nous pourrions aussi extraire dans les deux DataFrames les colonnes comprenant le mot Length et le mot Width, puis les fusionner.
iris_l <- iris[names(iris)[grepl("Length",names(iris), fixed = TRUE)]]
iris_w <- iris[names(iris)[grepl("Width",names(iris), fixed = TRUE)]]
iris_comb <- cbind(iris_l,iris_w)
names(iris_comb)## [1] "Sepal.Length" "Petal.Length" "Sepal.Width" "Petal.Width"1.4.2.9.2 Jointure de DataFrames
Une jointure est une opération un peu plus complexe qu’un simple ajout. L’idée est d’associer des informations de plusieurs DataFrames en utilisant une colonne (appelée une clef) présente dans les deux jeux de données. Nous distinguons plusieurs types de jointure :
- les jointures internes permettant de combiner les éléments communs entre deux DataFrames A et B;
- la jointure complète permettant de combiner les éléments présents dans A ou B;
- la jointure à gauche, permettant de ne conserver que les éléments présents dans A même s’ils n’ont pas de correspondance dans B.
Ces trois jointures sont présentées à la figure 1.17; pour ces trois cas, la colonne commune se nomme id.
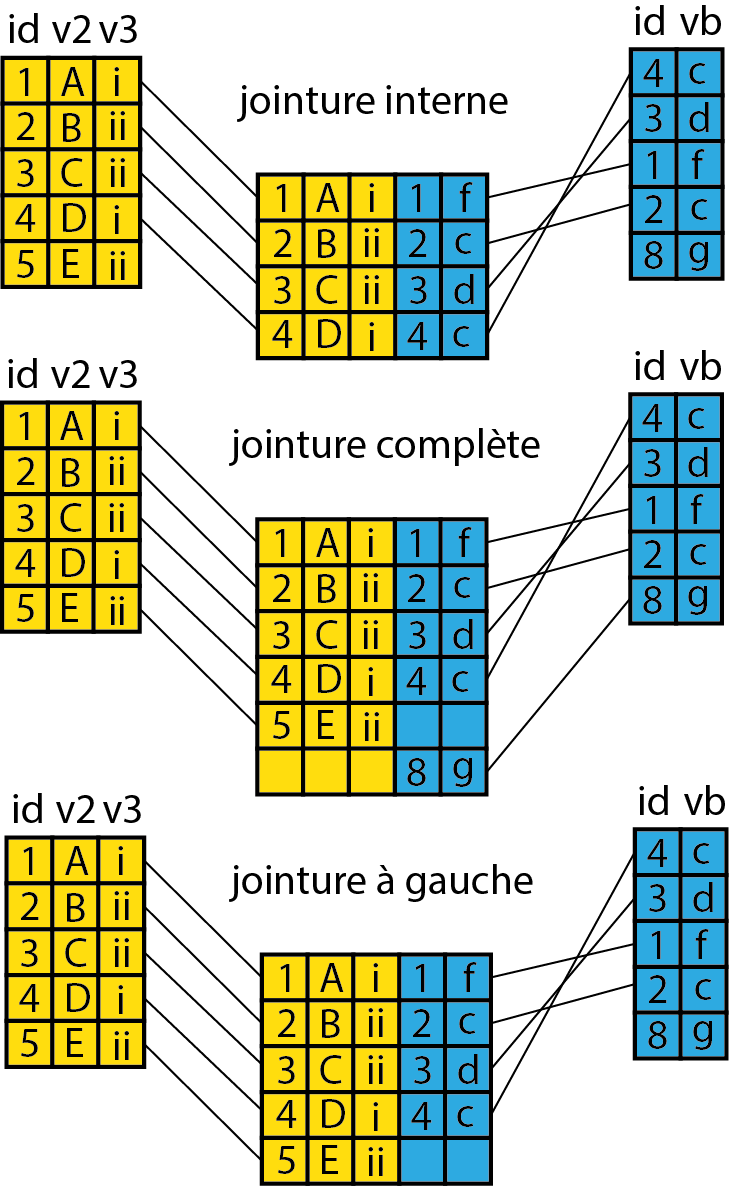
Figure 1.17: Jointure de DataFrames
Vous retiendrez que les deux dernières jointures peuvent produire des valeurs manquantes. Pour réaliser ces opérations, nous utilisons la fonction merge. Prenons un exemple simple à partir d’un petit jeu de données.
auteurs <- data.frame(
name = c("Tukey", "Venables", "Tierney", "Ripley", "McNeil", "Apparicio"),
nationality = c("US", "Australia", "US", "UK", "Australia", "Canada"),
retired = c("yes", rep("no", 5)))
livres <- data.frame(
aut = c("Tukey", "Venables", "Tierney", "Ripley", "Ripley", "McNeil","Wickham"),
title = c("Exploratory Data Analysis",
"Modern Applied Statistics ...",
"LISP-STAT",
"Spatial Statistics", "Stochastic Simulation",
"Interactive Data Analysis", "R for Data Science"))Nous avons donc deux DataFrames, le premier décrivant des auteurs et le second des livres. Effectuons une première jointure interne afin de savoir pour chaque livre la nationnalité de son auteur et si ce dernier est à la retraite.
df1 <- merge(livres, auteurs, #les deux DataFrames
by.x = "aut", by.y = 'name', #les noms des colonnes de jointures
all.x = FALSE, all.y = FALSE)
print(df1)## aut title nationality retired
## 1 McNeil Interactive Data Analysis Australia no
## 2 Ripley Spatial Statistics UK no
## 3 Ripley Stochastic Simulation UK no
## 4 Tierney LISP-STAT US no
## 5 Tukey Exploratory Data Analysis US yes
## 6 Venables Modern Applied Statistics ... Australia noCette jointure est interne, car les deux paramètres all.x et all.y ont pour valeur FALSE. Ainsi, nous indiquons à la fonction que nous ne souhaitons ni garder tous les éléments du premier DataFrame ni tous les éléments du second, mais uniquement les éléments présents dans les deux. Vous noterez ainsi que le livre “R for Data Science” n’est pas présent dans le jeu de données final, car son auteur “Wickham” ne fait pas partie du DataFrame auteurs. De même, l’auteur “Apparicio” n’apparaît pas dans la jointure, car aucun livre dans le DataFrame books n’a été écrit par cet auteur.
Pour conserver tous les livres, nous pouvons effectuer une jointure à gauche en renseignant all.x = TRUE. Nous forçons ainsi la fonction à garder tous les livres et à mettre des valeurs vides aux informations manquantes des auteurs.
df2 <- merge(livres, auteurs, #les deux DataFrames
by.x = "aut", by.y = 'name', #les noms des colonnes de jointures
all.x = TRUE, all.y = FALSE)
print(df2)## aut title nationality retired
## 1 McNeil Interactive Data Analysis Australia no
## 2 Ripley Spatial Statistics UK no
## 3 Ripley Stochastic Simulation UK no
## 4 Tierney LISP-STAT US no
## 5 Tukey Exploratory Data Analysis US yes
## 6 Venables Modern Applied Statistics ... Australia no
## 7 Wickham R for Data Science <NA> <NA>Pour garder tous les livres et tous les auteurs, nous pouvons faire une jointure complète en indiquant all.x = TRUE et all.y = TRUE.
df3 <- merge(livres, auteurs, #les deux DataFrames
by.x = "aut", by.y = 'name', #les noms des colonnes de jointures
all.x = TRUE, all.y = TRUE)
print(df3)## aut title nationality retired
## 1 Apparicio <NA> Canada no
## 2 McNeil Interactive Data Analysis Australia no
## 3 Ripley Spatial Statistics UK no
## 4 Ripley Stochastic Simulation UK no
## 5 Tierney LISP-STAT US no
## 6 Tukey Exploratory Data Analysis US yes
## 7 Venables Modern Applied Statistics ... Australia no
## 8 Wickham R for Data Science <NA> <NA>